说明
本篇文章使用例子来说明使用PyNE来进行基于网格的计算停机剂量率的流程。
本流程需要提前安装Trelis, DAGMC, ALARA和PyNE。
DAGMC的安装可以参考这里, PyNE的安装可以参考这里。ALARA的安装可以参考这里。
例子的重要性
使用的工具功能越多,那么使用起来一般就越复杂,新手就越难上手,新手总会在各种环节出现各种开发者意想不到的困难。即便软件官网上介绍了软件的各个工具的使用方法,但如果没有一个生动的例子来把这些应用工具的使用方法串起来的话,还是非常难的。
我自己作为一个PyNE系统的使用者,现在也参与了其中的部分开发工作,对此深有体会。对于新手的教学,告诉他们这个软件的各种功能是远远不够的,必须辅以实例,手把手的教会他们怎么用这个功能。用例子来说明这些问题是比较容易让新手接受的。我作为一个PyNE的使用者,也是一个新手。
这篇文章介绍了我如何从Trelis开始,一步一步实现计算一个简单模型的停机计量的过程的。
例题描述
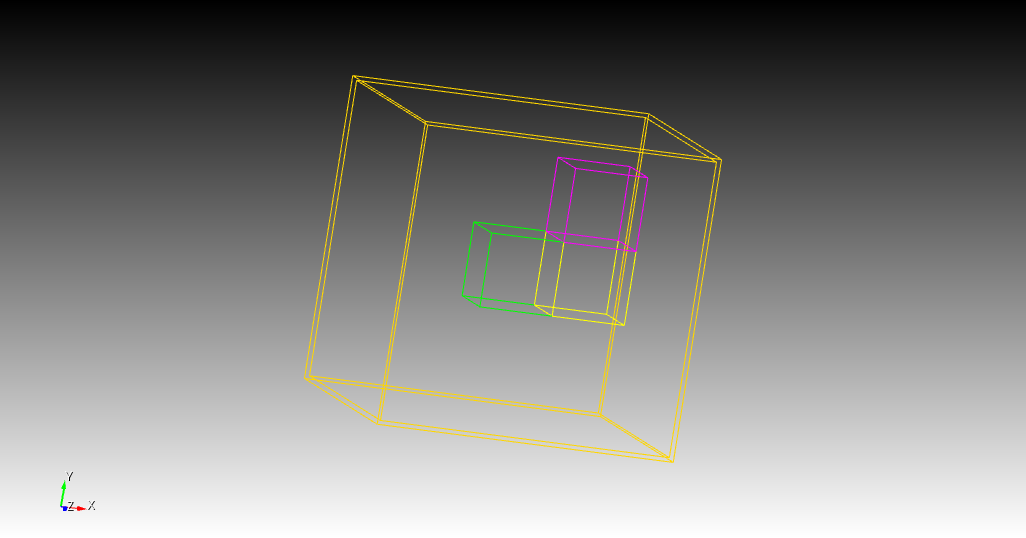
现有如下形状的几何体:
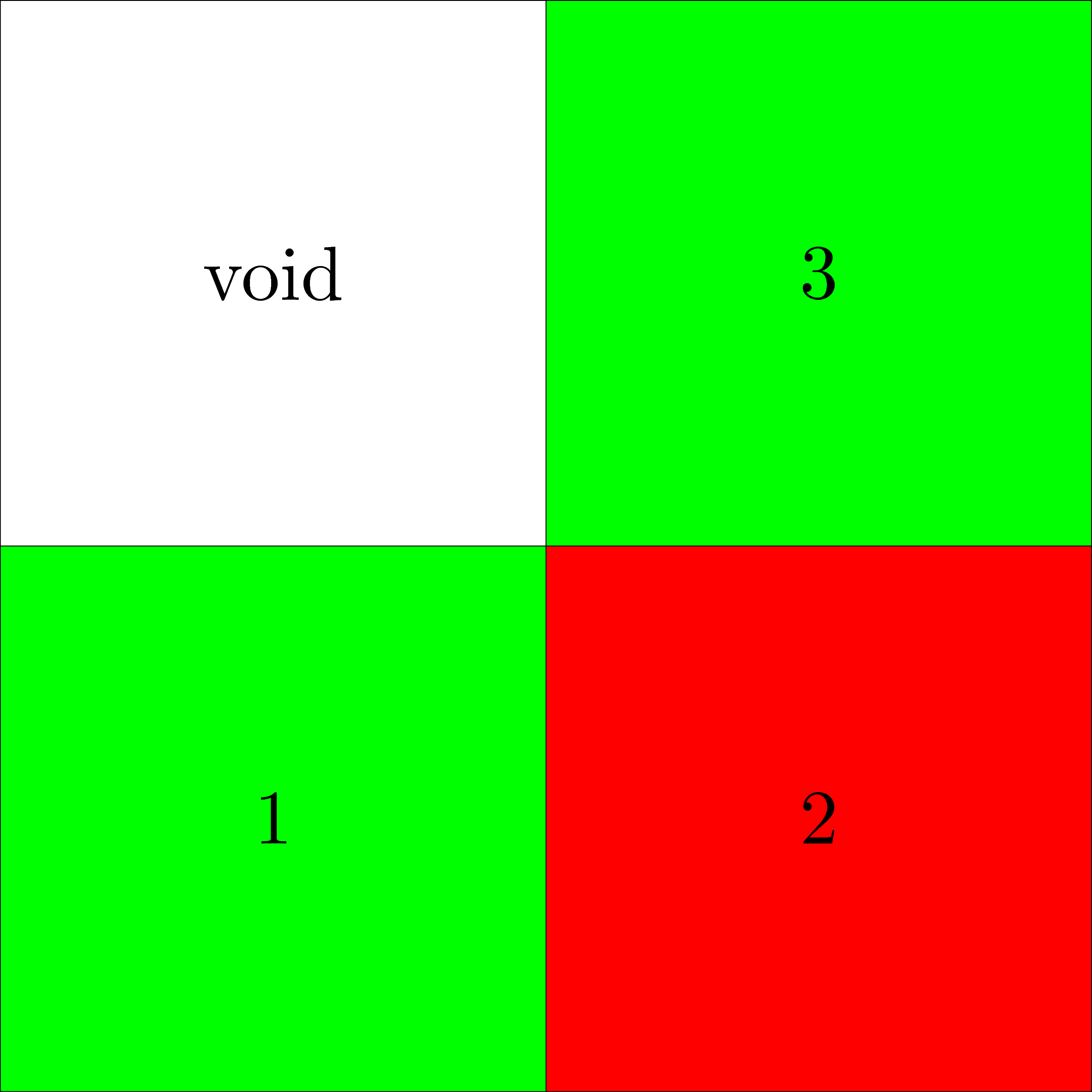
让点中子源以$10^{10}$个中子每秒的中子发射率辐照这几个栅元1年,然后停止辐照,考察停止辐照后1小时的栅元1, 2, 3的停机剂量率。
输入文件准备
要进行蒙特卡罗输运计算,我们需要提供以下信息:
- 几何信息,包括栅元和曲面。
- 材料信息。每个区域(栅元)是什么材料(材料的组成份额)
- 中子源信息。
- Tally信息。想统计的东西
- 其他信息。如截断卡,权重及权窗,计算终止条件等。
与普通MCNP计算不同,使用DAGMC计算时这些信息并不是全部都由一个输入文件提供,而是分别通过不同的文件提供。一个加载了几何和材料信息的h5m文件提供几何和材料信息。而其他信息则通过另一个input文本文件提供。
Trelis建模
几何体的建立
使用Trelis建立3个立方体,并在立方体外围设置一个graveyard区域,所有到达graveyard区域的中子将被杀死。graveyard的设置可以参考DAGMC关于graveyard的官方说明。
分组并赋材料
几何体设置好了之后,对这几个栅元进行分组并赋材料。
在trelis界面输入下面命令:
1 | trelis> group "mat:Water/rho:1.0" add vol 1 3 |
备注1:’trelis>’代表该命令是在trelis窗口中执行。
设置问题边界
边界包括终止边界和反射边界等。终止边界指的是我们不关心的区域,中子进入到终止边界后就会被杀死,这和MCNP中imp=0的区域相似。反射边界就是反射面,中子到达反射面后会被反射回去,在对称几何中很常用。
终止边界:graveyard
终止边界的设置实际上是通过对这个区域设置特殊材料实现的。比如此例题中需要将8号volume (可以简写为vol)设置为graveyard,可以使用下面命令进行设置:
1 | trelis> group "mat:Graveyard" add volume 8 |
反射边界
反射边界是通过指定反射面的方式来设置的。与volume类似,我们可以把一些面加入到一个group,然后将这个group设置为反射面即可。反射面属性的设置也是通过把group名字设置为特殊字符串的方式实现。
比如将10, 11号曲面设置为反射面,可以通过下面的命令:
1 | trelis> group "boundary:Reflecting" add surf 10 11 |
本例题中不需要将任何曲面设置为反射面。
imprint & merge
Imprint和merge是trelis中处理曲面的两个操作,他们的作用包括合并重合曲面和将无穷大的曲面根据几何体分割为总多的小的facet。功能说明可以参考trelis imprint and merge说明。
我们需要做的是,在trelis界面执行下面的操作:
1 | trelis> imprint body all |
导出几何文件
安装了DAGMC插件的trelis可以使用内置命令到处几何文件,使用方法可以通过下面命令查看帮助信息:
1 | trelis> help dagmc |
也可以参考官方文档。
对于本例题,我们可以在trelis中执行下面命令将几何信息存放到”geometry1.h5m”中:
1 | trelis> export dagmc geometry1.h5m |
至此,我们完成了几何文件的制作。
注意:在没有设置trelis默认导出文件路径的情况下,此时生成的”geometry1.h5m”文件并不一定在当前文件夹,可能存放于$HOME/Documents中,也可能在其他常用默认文件夹中
制作材料
我们需要首先制作本例题需要使用的材料库,包括Water和Steel两种材料。可以参考PyNE材料库制作。
下面是制作本例子中的”Water”和”Steel”用到的python脚本文件:
1 | #!/usr/bin/python |
备注:制作材料的时候会调用PyNE,需要保证此时系统LD_LIBRARY_PATH中的MOAB是4.9.1版本的。
整合几何与材料
按照UWUW流程制作材料,使用UWUW_preprocessor:
1 | uwuw_preproc <dagmc h5m filename> -v -l <path to nuclear data library> |
假设我们制作了材料库并存放在”material_lib1.h5”中,我们接下来就可以执行:
1 | uwuw_preproc geometry1.h5m -v -l material_lib1.h5 |
DAGMC input文件准备
DAGMC的input文件中需要给定普通MCNP中除了栅元卡和曲面卡以及材料卡的其他部分,包括但不限于:
- 中子源定义,必要项
- 计算终止条件,必要项
- Tally卡,可选项,但一般都会有
- 其他功能性辅助卡,可选项,根据实际情况添加
对于此例子的一个简单input文件如下:
1 | C test1 input |
中子输运计算
使用DAGMC进行输运计算时,需要通过i=**来指定输入文件,通过g=**来指定加载了材料的几何文件,详细运行指令可以参考官方文档。
对于本例题,可以使用下面的方法运行本例子。
单线程运行:
1 | # 需要先保障系统能够找到正确的mcnp5.mpi,找不到的话需要给出完整路径 |
多线程运行:
1 | mpirun -np n mcnp5.mpi i=input g=geom.h5m |
备注:input文件和几何文件的名称长度都不能超过8个字符,可以使用ln -sf original_long_name.h5m geom.h5m把原文件链接到一个较短名字的文件后再运行。
计算完成之后,我们便得到了一个meshtal文件。
活化计算
中子输运完成后,我们开始正式调用PyNE的R2S计算模块的功能进行停机剂量率计算了。
进行停机剂量率计算,我们需要的主要信息包括:
- 每个网格单元的材料成分。必须项。由同时包含了几何和材料的geom.h5m文件提供。(此文件就是进行DAGMC计算的那个)
- 每个网格单元的中子通量能谱。必须项。由meshtal文件提供。
- 活化计算能力。必须项。由Analytic and Laplacian Adaptive Radioactivity Analysis软件:ALARA提供。
- 辐照及冷却方案。必须项。需要在R2S过程中指定。
在我们的工作文件夹下新建一个子文件夹用来存放R2S计算相关的文件。
1 | mkdir r2s_run |
初始化配置:setup
初始化配置r2s。执行:
1 | r2s.py setup |
执行之后我们应该能够看到下面的信息:
1 | # 一些warning |
修改配置文件
setup这一步会生成两个文件:config.ini和alara_params.txt,我们需要修改这两个文件中的一些设置,包括辐照方案的设置和一些计算细节的设置。
修改config.ini设置
一般情况下,我们需要注意的几个设置是:
- structured: 是否是结构化网格。结构化网格就是指MCNP中用的xyz或cyl网格。我们应该选True
- sub_voxel: 是否采用sub_voxel模式,sub_voxel模式是我正在添加的代码,还没有完成,暂时不能选用,因此是False
- reverse: 反转中子通量顺序。DAGMC输出的中子通量是按照从低能到高能排序,而用ALARA进行活化计算时需要按照从高能到低能读取,因此需要把中子通量逆序排列。这里填True。
1 | [general] |
修改alara_params.txt
1 | material_lib alara_matlib |
r2s.py step1
r2s.py step1是用来根据r2s.py setup生成的配置文件生成ALARA输入文件,中子通量文件和ALARA材料库文件的。
当我们修改好上面两个文件后,就可以使用r2s.py生成ALARA计算需要的文件了:
1 | r2s.py step1 |
材料数据库准备
ALARA输入文件中有三行是材料数据库相关的信息。
- material_lib alara_matlib 这个alara_matlib是alara需要读取的材料成功定义的文件名。这个文件是会由程序从test1_geometry.h5m (geom.h5m)中读取的,不需要做修改。
- element_lib data/nuclib 这个文件是定义元素的丰度用的。默认是自然丰度,也可以自己定义修改丰度。自己定义一个元素丰度数据表显然是非常麻烦的,在ALARA的安装文件中$ALARA/data下有一个elelib.std,可以把这个文件拷贝到工作文件夹下并重命名为nucib即可(也可以设置element_lib elelib.std)。但是这个elelib.std只能用于材料定义是按照元素定义的,如果有针对核素的定义,则这个就不能用了。需要将元素扩展成核素,需要后续介的方法单独处理。
- data_library alaralib truncated_fendl2bin (ALARA应该配备fendl3bin)但是并没有,ALARA安装文件中的sample/datam文件夹下只有truncated_fendl2bin数据库。我们可以把这个数据库相关的文件拷贝到工作文件夹。
当材料定义是针对核素定义了的化,普通的elelib.std就没办法使用了,会出现错误:
1 | 310: Could not find element <string> in element library. |
alara计算
当step1完成后,程序会给出运行alara的提示,按照提示运行:
1 | alara alara_inp > output.txt |
等待alara运行完成,会生成几个文件:
- output.txt: ALARA输出文件,里面有活化后材料的核素组成以及光子发射率数据
- phtn_src: ALARA输出文件,里面有详细的各个各个材料各个核素缠身的各个能群的光子数据
r2s step2
ALARA计算完成后,运行:
1 | r2s.py step2 |
执行完成后,我们可以看到工作文件夹中多了几个文件:
- phtn_src.h5: HDF5格式的光子源数据,无法直接打开读取,只能程序读。
- source_i.h5m: 第i个冷却时间点的网格光子源数据。
- total_photon_source_intensites.txt: 里面是各个冷却时间节点的总光子发射率。
你
光子输运
获得光子源后,我们就可以进行光子输运计算了。计算前必须确认DAGMC是已经包含了源子程序的版本,如果没有,需要先参考编译带用于R2S的源子程序的DAGMC安装DAGMC。
文件准备
进行光子输运计算,我们需要提供几何文件,材料,光子源,以及需要统计的信息。因此需要准备的文件有:
- geom.h5m: 就是中子输运时用的那个文件,即本例子中的test1_geometry.h5m,它包含了几何和材料信息
- source.h5m:光子源信息文件,源子程序需要从这里读取信息。由于MCNP无法从输入文件中传递字符串,所以这个文件名称不能改变,必须是source.h5m。
- input: 提供tally信息,另外需要在这个文件中定义idum,用于控制源子程序过程。
准备好这些文件后,我们还需要对这些文件进行一些小修改。
- input文件修改:首先,需要加上mode p, 然后修改tally部分。
- input文件修改:注释掉原本的SDEF卡
我们还需要修改栅元的重要性,因为中子输运时的文件里面的重要性是默认为中子的重要性,没有光子的重要性。但是这个重要性信息是写在h5m文件里面的,没法直接修改。
我们需要先运行一次这个文件:
1 | mcnp5.mpi i=input g=geom.h5m |
现在这个运行肯定会出现错误,说粒子产生子重要性为0的区域。但是同时这个过程会生成一个可编辑的lcad文件,我们可以修改这个lcad文件,在重要性部分添加上imp:p=1/imp:p=0,将这个文件重命名为lcad_m,删掉过程文件fcad, outp, runtpe后再次运行
1 | mcnp5.mpi i=input g=geom.h5m l=lcad_m |
之后就得到结果,至此停机剂量的计算完成。
备注:在计算得到结果之后,还可以把几何图形及计算结果的网格数据进行可视化绘图。绘图方法可以参考DAGMC数据绘图。